| 研究期間 | 2020/4/1 - 2022/9/30 |
| 研究課題名 | 【終了】アトピー性疾患の新規治療ターゲットの解明 |
| カテゴリー | 全てのクラスター、研究クラスター一覧、登録クラスター、医学、終了した研究クラスター、選定クラスター、基礎、臨床、薬学、創薬、バイオ、生物、ゲノム、バイオ |
| SDGs | 9.イノベーション |
| 応募課題 | |
| クラスター長氏名 | 峯岸 克行(先端酵素学研究所、免疫アレルギー学分野、教授) |
| 所属する研究者氏名 | 竹本 龍也 先端酵素学研究所 発生生物学分野 教授 |
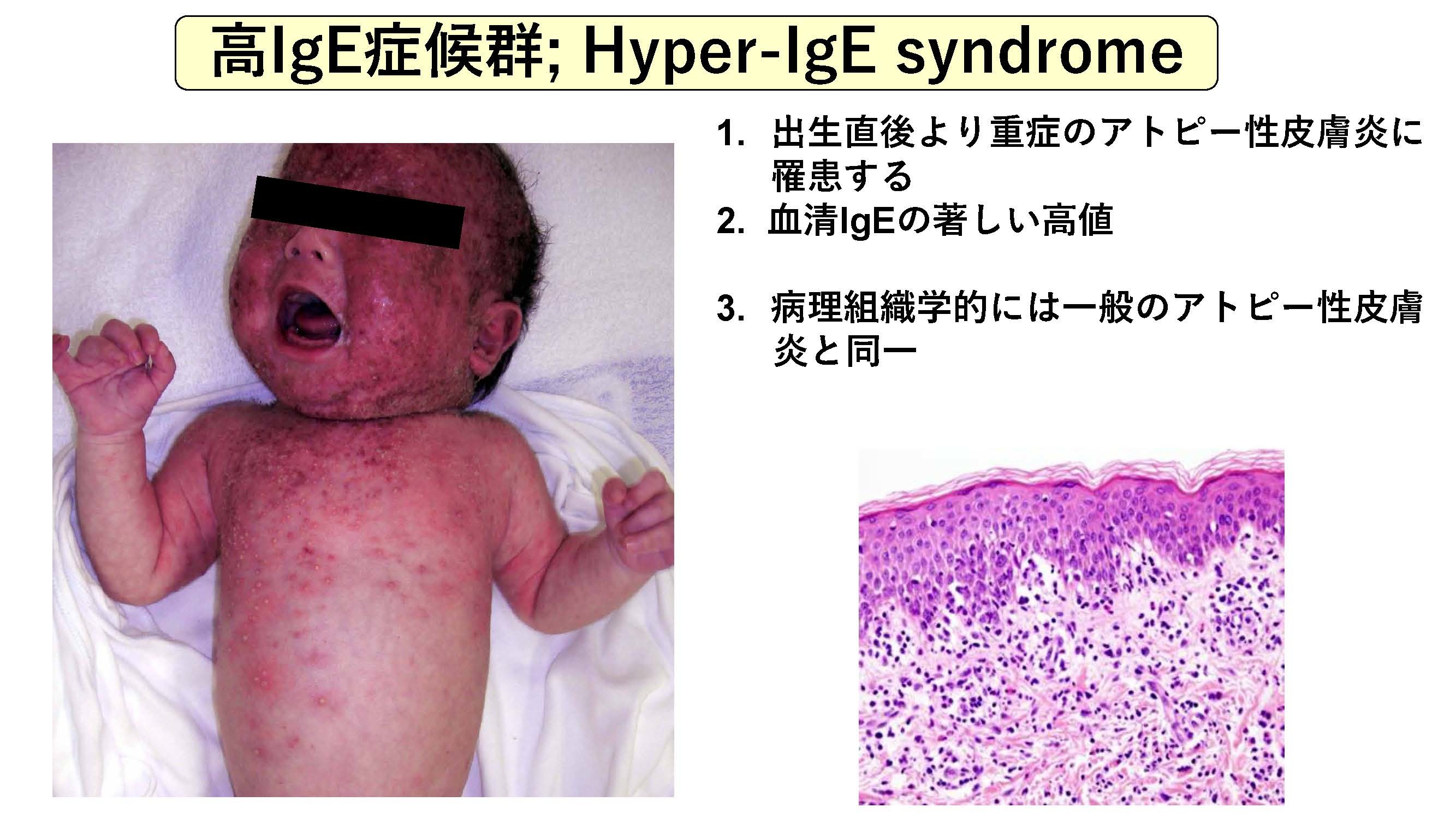
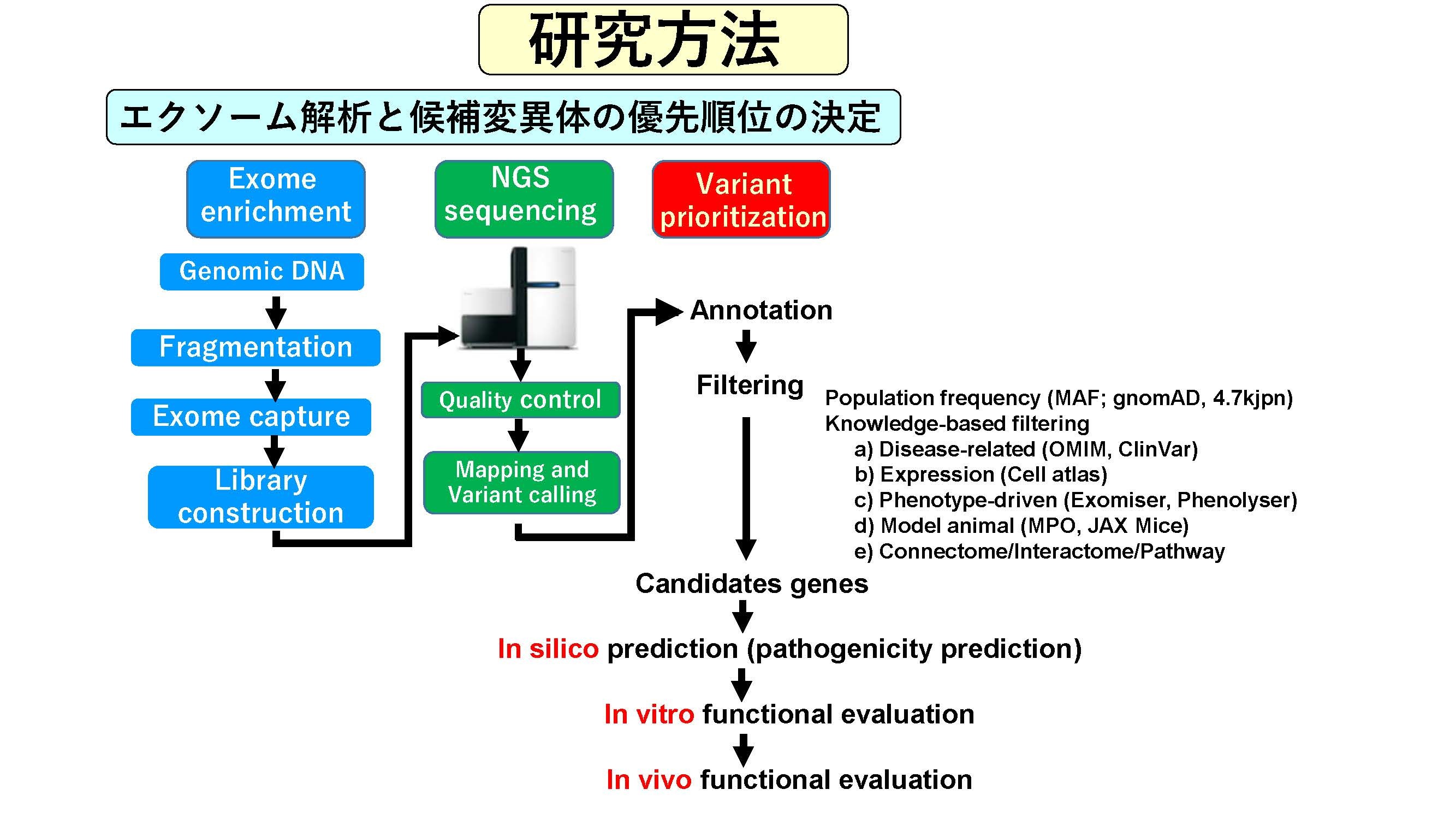
| 研究概要 | 【Introduction】 本研究提案では、人類の健康と福祉を増進する画期的な医療手段を提供することを最終目的とする。そのために研究成果を論文発表し、さらにその研究成果を社会に還元する手段として、医薬品として開発することを目指す。本研究では特に、日本人の約25%が罹患するアトピー性皮膚炎、気管支喘息、アトピー性鼻炎等の血清IgEの上昇を伴うアトピー性疾患を研究対象とし、新規の治療法の開発を目的とした研究を展開する。この実現に向けて、発生生物学を専門とする先端酵素学研究所の竹本教授と免疫アレルギー学を専門とする本研究クラスターの研究代表者が研究分野を超えた異分野融合の共同研究を実施し、世界的に多くの患者を苦しめているアトピー性疾患の新規治療ターゲットの発見を目的とした研究を実施する。 アトピー性疾患はアレルゲンがヒトの上皮組織に侵入し、上皮の樹状細胞に貪食されることを契機に発症する(研究概要図 画像1参照)。アレルゲンを貪食した樹状細胞はその所属リンパ節に移動し、T細胞と相互作用することにより複雑な免疫応答が開始され、最終的にアレルゲンに特異的なIgEクラスの抗体が産生される。IgEはマスト細胞と好塩基球の細胞表面に存在する高親和性IgEレセプターに結合し、再度侵入してきたアレルゲンに架橋されることにより脱顆粒し、各種のケミカルメディエーターとサイトカイン・ケモカイン等が放出される。さらに好酸球が動員されてアレルギー性の慢性炎症が発症する。これまでの研究により上皮性サイトカインのIL-25/IL-33/TSLP, Th2サイトカインのIL-4/IL-13、IgE、各種ケミカルメディエーター、好酸球/IL-5等が治療ターゲットとして同定され治療薬の開発が進んでいる。しかし、樹状細胞からT細胞とB細胞を介してIgEが産生されるプロセスに関して、特にIgE産生制御のメカニズムが十分に理解されていないため、この領域でのアトピーに対する有効な治療ターゲットは見いだされていない。 この課題を解決する目的で、研究代表者らは重症のアトピー性疾患を合併する免疫難病、高IgE症候群に着目した(画像2)。高IgE症候群は、全例で重症のアトピー性皮膚炎、血清IgEの著しい高値を呈する原因不明の免疫難病であった。その主要な原因がSTAT3のドミナントネガティブ(DN)変異であることを研究代表者らが世界で初めて明らかにし、その後の検討により高IgE症候群の約3分の2はSTAT3の遺伝子異常が原因で発症することが明らかになったが、残りの約3分の1は未だその原因がほとんど明らかになっていない。本研究提案では、原因が不明の高IgE症候群に注目し、次世代シークエンサーによるエクソーム解析を実施する。エクソーム解析で見出した候補変異体をin silicoで詳細に解析し、さらに候補変異体のin vitro機能解析を網羅的に実施する。エクソーム解析に候補変異体の網羅的なin vitro機能解析を組み合わせる研究手法は、高IgE症候群の病因研究においては世界的にも全く行われておらず、研究の新規性・先進性・優位性が高い。そこで異常を認めたものに対して、疾患モデルマウスをCRISPR/Cas9を用いたゲノム編集により作成し、エクソーム解析で見出した遺伝子変異が疾患発症の原因であることをin vivoで証明する。この研究方法の有効性を検討するための予備実験として、in silicoとin vitroの機能解析で見出した4個の候補変異体を発現するモデルマウスを作成したところ、そのうちの1系統で著しい高IgE血症とアトピー症状の増悪を認めた。現在この分子がアトピーの新規治療標的になる可能性を製薬会社と共同で検討を開始している。 【研究方法】 高IgE症候群の新規原因遺伝子を発見するために、これまでに報告されている既知の原因遺伝子に異常のない高IgE症候群症例のゲノムDNAを対象として次世代シークエンサーによる検討を行う(画像3)。患児の末梢血由来ゲノムDNAからエクソン領域を濃縮し、エクソーム解析を実施する。得られたデータを次世代シークエンサーの解析パイプラインで検討し、アノテーションを実施する。原因遺伝子の探索は、遺伝形式を仮定して、それぞれの仮説と一致したホモ、ヘテロ、コンパウンドヘテロの変異を抽出したのち、それが原因遺伝子変異である可能性をバイオインフォーマティクスにより検討する。具体的には、それぞれの候補遺伝子に対して、正常人集団中での多型頻度(population frequency)をgnomADと4.7kjpnデータベースにより評価する。疾患・表現型との関連、免疫系での発現やモデル動物での表現系、パスウェイ解析、機械学習による変異体の病原性の予測等を実施し、候補遺伝子変異に優先順位をつけ、優先順位の高い候補遺伝子変異体のin vitro機能解析を研究代表者の実施する。 これまでの予備的検討により、候補遺伝子変異体のほとんどは、その遺伝子変異の機能的影響が予測可能なナンセンス変異、フレームシフト変異、スプライシング変異等ではなく、ほとんどがミスセンス変異体であることが明らかになっている。さらに、高IgE症候群患児に特異的な特定の遺伝子変異の集積や、特定の遺伝子への変異の集積がないことが明らかになっており、候補変異体が3000個ほど存在するためin vivo機能解析は困難で、in vitro機能解析は必須である。 候補変異体と野生型の遺伝子を発現ベクターにクローニングし、HEK293T細胞、Jurkat細胞等に発現し、変異体発現の影響をレポーターアッセイ、ウエスタンブロッティング、フローサイトメトリー、定量PCR等の各種検査法にて野生型発現と比較検討する。試験管内で実際に機能異常を有する変異体を選択し、疾患の原因である可能性により優先順位をつける。 次に先端酵素学研究所・発生生物学分野の竹本教授の研究室においてin vitro機能解析で選択した候補変異体のゲノム編集モデルマウスを作成する。これまでの予備的検討により、候補変異体のほとんどがミスセンス変異体で、特定の遺伝子変異の集積や、特定の遺伝子への変異の集積等がないことが明らかになっているため、このin vivo機能解析は必須である。マウスの受精卵にCas9 タンパク質、ガイドRNA、1本鎖のドナーDNAをエレクトロポレーションし、部位特異的なDNA2本鎖切断を誘導しノックインマウスを作成する。竹本教授の研究室においては、CRISPR/Cas9による多数の疾患モデルマウス作成しており、確実に短期間でモデルマウスを得ることができる。得られたゲノム編集マウスを研究代表者の研究室にて詳細に解析する。その際に高IgE症候群症例の臨床情報を参考にして、血清のIgE/IgG1値等の免疫グロブリン値、免疫した際の抗原特異的IgE/IgG1産生能、アレルギー性炎症を誘導した際の炎症の増悪の有無等でまずスクリーニングを実施する。 【準備状況】 これまでに、高IgE症候群100症例の次世代シークエンサーによるエクソーム解析とin silicoの解析が終了している。さらに、これにより見出した3000個の候補変異体の1次の in vitro機能解析が終了している。4個の候補変異体のin vivo機能解析が終了し、1個が新規の高IgE症候群の原因遺伝子であることが明らかになっている。見出した新規の治療ターゲットに興味を持つ2社の製薬企業と秘密保持契約を締結の上で共同研究の準備を開始している。 |
| 研究概要図 | |
| 研究者の役割分担 | 研究代表者 高IgE症候群のエクソーム解析 エクソーム変異体のin silico解析 エクソーム変異体のin vitro解析 疾患モデルマウスの免疫能解析 竹本 教授 疾患モデルマウスの樹立 |
徳島大学の研究特集